- BIOLOGIE MOLÉCULAIRE
- BIOLOGIE MOLÉCULAIREFormés de molécules, les êtres vivants (qu’ils soient uni- ou pluricellulaires) sont des systèmes organisés, en perpétuel état de renouvellement. Échangeant à tout instant avec le milieu dans lequel ils se trouvent des flux d’information, de matière et d’énergie, ils doivent de conserver leurs structures, de maintenir leur individualité, au fait qu’ils sont dotés d’un répertoire d’instructions, d’un programme génétique, qui est propre à chacun d’eux.Les êtres vivants sont aussi, de tous les systèmes que nous connaissons, les seuls qui aient la capacité de se reproduire semblables à eux-mêmes, de transmettre leurs caractères à leur descendance, de leur transmettre également les modifications de leur patrimoine génétique, d’évoluer. Là encore, c’est le programme génétique qui est maître d’œuvre de même qu’il l’est pour le déroulement des processus qui, de l’œuf ou de la graine, aboutissent à la formation d’un organisme, à la mise en place des processus de régulation nécessaires à son fonctionnement, aux traits de comportement qui lui permettent de s’adapter aux conditions changeantes du milieu comme de conduire à la reproduction sexuée et même à la formation de certaines communautés.La biologie moléculaire est essentiellement l’étude des molécules qui constituent les êtres vivants et des processus moléculaires qui assurent leur fonctionnement. Ces molécules ne sont pas spécifiques d’une certaine forme de matière qui serait la matière vivante: il n’y a pas de matière vivante, pas de molécules vivantes... il y a des êtres vivants. On trouve cependant chez ces derniers des espèces moléculaires que l’on ne trouve pas dans le monde inanimé tel que nous le connaissons actuellement, en particulier des assemblages, des macromolécules: acides nucléiques, protéines, polysaccharides... qui participent à la construction des cellules, des organes et en assurent les fonctions.Ce sont les structures et les propriétés fonctionnelles de ces molécules, leur synthèse et leur dégradation, les relations qu’elles entretiennent entre elles, les systèmes de régulation mis en jeu qui intéressent le biologiste moléculaire. C’est, en effet, de leur connaissance que dépend au premier chef la compréhension des caractères propres aux êtres vivants. En ce sens, l’approche moléculaire ne constitue pas seulement un aboutissement dans l’approfondissement des connaissances mais aussi, et surtout, une façon nouvelle d’aborder les grands problèmes de la biologie. Ce faisant, le biologiste n’oublie pas que la plus petite unité qui manifeste ces caractères est la cellule. Il n’oublie pas non plus qu’une cellule n’est pas la somme de ses parties, que c’est un ensemble où structures et fonctions sont intégrées, et qu’il en est de même pour les autres niveaux d’organisation: la connaissance des propriétés des sous-ensembles ne suffit pas pour comprendre les caractéristiques de l’ensemble: celui-ci manifeste de nouvelles propriétés par rapport à celles des éléments dont il est formé. Mais, dans sa démarche, le biologiste moléculaire parvient à mettre à jour ce qui est commun à tous les êtres vivants et qui se trouve masqué par l’extraordinaire diversité des «solutions» adoptées au cours de l’évolution pour résoudre un même problème: subsister et transmettre.Sous-jacentes, en effet, à cette extraordinaire variabilité qui caractérise les populations naturelles, la génétique, la biochimie et, produit de leur fusion, la génétique ou biologie moléculaire, découvrent une profonde unité: unité de composition, unité de structure, unité de fonctionnement. Ce sont chez tous les organismes, comme chez tous les virus, les mêmes quatre bases nucléiques, adénine, guanine, thymine, cytosine, qui forment les acides désoxyribonucléiques (ADN), dont les segments constituent autant de gènes définis par l’ordre dans lequel ces bases y sont disposées. Ce sont chez tous les êtres vivants (à quelques détails près) les mêmes systèmes qui assurent, tout d’abord, la transcription puis la traduction des messages génétiques, aboutissant à la synthèse de protéines qui, chez tous les êtres vivants, sont formées à partir des mêmes 20 acides aminés principaux; le dictionnaire des groupes de 3 bases (codons) qui spécifient la mise en place des acides aminés, étant le même (à quelques rares exceptions près) chez tous les êtres vivants. C’est cette unité de structure et de fonctionnement qui permet, ayant isolé un gène d’un organisme donné, de l’incorporer au matériel génétique d’un autre organisme, et c’est cela qui fonde le génie génétique.Enfin, quand il y a reproduction sexuée, ce sont les mêmes lois qui président à la distribution et à la transmission des gènes.Unité et diversité ont essentiellement pour origine le mode de réplication dit semi-conservatif de l’ADN qui, d’une part, assure la reproduction conforme du matériel héréditaire, d’autre part, assure que toute modification de ce matériel sera transmise à la descendance, apparaissant comme une mutation sur laquelle joueront les processus de sélection. Toutefois, l’acide désoxyribonucléique (ADN) constitue le matériel héréditaire de tous les organismes, mais non celui de certains virus, où la même fonction est assurée par l’acide ribonucléique (ARN) sans que cela porte atteinte néanmoins aux lois fondamentales de l’information génétique [cf. GÉNOME].Nous savons donc à présent ce qu’est l’information génétique, comment elle est transmise de génération en génération, nous savons comment cette information peut être modifiée, comment elle est déchiffrée, comment elle conditionne la structure et le fonctionnement des êtres vivants. Et l’essentiel peut être résumé sous une forme qui eût ravi Pythagore:
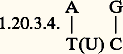 la mise en place d’1 acide aminé donné parmi les 20 qui composent les protéines est sous la dépendance d’un groupe de 3 bases parmi les 4 qui composent l’ADN, la spécificité étant assurée par les liaisons qui s’établissent entre les bases complémentaires adénine (A) et thymine (T) ou uracile (U), et guanine (G) et cytosine (C).C’est à l’ensemble des travaux qui portent sur la structure, le mode d’expression et le mode de reproduction des gènes qu’est souvent réservé, dans un sens plus restreint, le terme de biologie moléculaire. L’essentiel en est exposé ci-dessous.1. Bases de la biologie moléculaireL’acide désoxyribonucléique est fait de l’assemblage de motifs nucléotidiques , constitués par l’union d’un sucre, le désoxyribose, et d’une base nucléique (A, T, G ou C, c’est-à-dire adénine, thymine, guanine ou cytosine). Ces nucléotides sont reliés par l’intermédiaire d’un chaînon phosphoryle (P dans la figure 1 a).La chaîne phosphonucléotidique ainsi définie représente l’un des deux brins dont la molécule d’ADN est constituée: autrement dit, celle-ci est bicaténaire , avec deux brins accolés longitudinalement par des liaisons de faible énergie établies entre A et T, d’une part, C et G, d’autre part (fig. 1b). La complémentarité réciproque des deux brins conditionne donc la stabilité de cette structure. Dans ce cas, la molécule bicaténaire s’enroule en double hélice (fig. 7). Le fait que chacun des brins soit complémentaire de l’autre est dû aux modalités de la réplication de l’ADN.La bipartition qui va s’opérer à partir de la cellule mère, pour donner naissance à deux cellules filles, est précédée par la construction de constituants cellulaires qui seront donc partagés entre les cellules nouvelles. Dans le cas de l’ADN, chaque brin va servir de matrice pour la synthèse d’une chaîne complémentaire, qui en est donc la réplique . Ainsi se forme une nouvelle molécule (fig. 1c) dans laquelle l’un des brins est hérité de la molécule affectée par la réplication: celle-ci est donc semi-conservatrice. C’est pourquoi les cellules filles reçoivent l’une et l’autre des molécules d’ADN absolument identiques (à cette réserve près que le brin «ancien» de chacune d’elles n’est pas le même). Lorsque deux cellules filles ont reçu identiquement le même matériel génétique, elles synthétisent les mêmes protéines, et c’est pourquoi elles ont les mêmes structures et accomplissent les mêmes réactions chimiques.Mais, au contraire, en cas de mutation (fig. 1d), une partie du message se trouvant modifiée, la protéine régie par la séquence en cause sera différente, ce qui aura pour conséquence d’imposer un caractère nouveau à l’organisme et à ses descendants, à moins qu’une mutation «reverse» ne rétablisse l’ancien état des choses.Pour expliquer que la biosynthèse des protéines soit ainsi spécifiquement conjuguée à celles des acides nucléiques, il faut partir du fait que toute protéine est formée de chaînes d’acides aminés. Ceux-ci diffèrent par un radical R. Il en existe vingt espèces chimiques différentes selon les particularités dudit radical (R1...R20). La protéine renferme donc une séquence plus ou moins longue (une centaine à plusieurs centaines) d’acides aminés.Une fois synthétisée, ou au cours de sa synthèse, chacune de ces longues chaînes se replie sur elle-même, la structure tridimensionnelle adoptée ne dépendant que de la géométrie des atomes et étant stabilisée grâce aux liaisons hydrogènes: cette structure tridimensionnelle impose à la protéine sa fonction.Or l’ordre et la nature des acides aminés mis en place est sous la dépendance de l’ordre et de la nature des bases de l’ADN. Tout se passe comme s’il fallait traduire un alphabet à 4 lettres en un alphabet à 20 lettres afin que la coïncidence des deux séquences, nucléique et protéique, soit possible. L’expression génétique comprend ainsi deux étapes (fig. 2).Il y a d’abord une étape de transcription au cours de laquelle l’un des brins de l’ADN (tantôt l’un, tantôt l’autre, mais jamais les deux ensemble) sert de matrice pour la synthèse d’une chaîne complémentaire d’acide ribonucléique. Seules différences avec l’ADN: le sucre est ici le ribose et la thymine est remplacée par l’uracile. L’acide ribonucléique destiné à la biosynthèse des protéines est dit ARN messager (ARNm).Puis, une étape de traduction fait intervenir de nombreuses enzymes et de nombreux facteurs:– les ARN de transfert qui peuvent servir de «traducteurs» du fait qu’ils «connaissent» les deux langages, le langage nucléique et le langage protéique; un ARN de transfert donné peut en effet se lier de façon spécifique à un acide aminé donné (réaction catalysée par une enzyme spécifique); ce même ARN de transfert possède un groupe de trois bases (anti-codon) qui sont complémentaires d’un groupe de trois bases (codon) de l’ARN messager (fig. 3);– Les ribosomes, particules de nature ribonucléoprotéique, où se positionnent l’ARN messager et les ARN de transfert liés à leurs acides aminés, ce qui permet les liaisons entre ces acides aminés, les ARN de transfert étant détachés du complexe au fur et à mesure que chaque liaison s’effectue.La traduction s’effectue donc séquentiellement, à partir d’un point de départ et toujours dans la même direction, chaque acide aminé étant mis en place selon l’ordre qui lui est dicté par les séquences de codons de l’ARN messager qui dérive directement de la séquence des bases du segment de l’ADN du gène impliqué.La conception des mécanismes d’expression des gènes, qui vient d’être exposée, est issue de l’étude des bactéries et des virus, notamment les bactériophages. Les grandes étapes de ce processus, transcription et traduction, ont été retrouvées chez les organismes qui possèdent un véritable noyau, distinct du cytoplasme (les organismes eucaryotes) mais les modalités selon lesquelles ces processus s’y effectuent sont différentes, particulièrement chez les organismes pluricellulaires.Les différences portent sur plusieurs points, dont les principaux sont les suivants:– au contraire de ce qui se passe chez les bactéries, où la traduction du message (fig. 4) s’effectue dès qu’il commence à être synthétisé, dans les cellules eucaryotes le produit de transcription n’est pas immédiatement traduit, ce qui implique une certaine stabilité des ARN messagers;– le produit primaire de la transcription, qui se trouve dans le noyau cellulaire, est souvent (mais pas toujours) une molécule d’ARN géante comportant des séquences de bases qui ne seront pas traduites et qui est profondément remaniée avant d’aboutir à l’ARN messager proprement dit, selon le schéma de la figure 5.L’étude de molécules d’ARN intermédiaires entre le produit initial de la transcription de ces gènes dits «éclatés» ou «en mosaïque» et l’ARN messager, et qui permettent de saisir les étapes de la maturation, révèle que excisions et liaisons ne se font pas obligatoirement dans l’ordre où se situent les différentes séquences.Des séquences de bases (introns) n’auront pas codé pour l’ARN messager, elles n’en ont pas moins un rôle important. Il semble, en effet, que certaines d’entre elles soient nécessaires aux processus qui aboutissent à la molécule finale. C’est ainsi qu’une mutation intéressant l’un des introns du gène éclaté qui spécifie la synthèse de la chaîne 廓 de la globine humaine (et participe à l’édification de l’hémoglobine) provoque la formation d’ARN messager dont les propriétés ne sont pas normales. Le défaut dans la biosynthèse de la chaîne 廓 qui s’ensuit, entraîne une forme héréditaire de 廓 thalassémie, anémie grave.Il doit donc s’exercer au niveau des processus de maturation des ARN messagers des régulations dont les mécanismes sont actuellement à l’étude. Quoi qu’il en soit, les réactions mises en jeu dans l’excision des introns et l’aboutage des exons, sont d’une extrême précision. Un exemple en est fourni par le gène de l’insuline humaine (fig. 6).2. HistoriqueOn tentera ici, sans vouloir être exhaustif, de dégager les raisons des progrès très rapides qu’a connus et que connaît la biologie moléculaire en s’attachant plus particulièrement à ce qui touche l’étude de la structure et du mode de fonctionnement des gènes.L’hypothèse un gène (= acide nucléique) = une enzyme (= protéine)Tout d’abord, le problème était clairement posé. Dans les années cinquante, on savait déjà grâce aux travaux d’Ephrussi et Beadle (1935-1940) sur la Drosophile, de Beadle et Tatum (1940-1945) sur le champignon Neurospora que chaque réaction biochimique est sous le contrôle d’un gène donné et que ce contrôle s’exerce sur la protéine enzymatique qui catalyse la réaction. Ainsi se trouvaient confirmées les idées émises par Cuénot, d’une part, par Garrod, d’autre part, dès les premières années du siècle.Par ailleurs, Avery, Mac Leod et Mc Carty montraient en 1944 que le facteur mis en évidence par Griffith (1928) qui, extrait de pneumocoques virulents, confère de façon héréditaire cette virulence à des pneumocoques non virulents, est de l’ADN. Tout se passant comme si le ou les gènes gouvernant la biosynthèse de la capsule polysaccharidique des pneumocoques virulents avaient été transférés aux pneumocoques dépourvus de toute capsule et s’y exprimaient, ce qui signifiait que le gène bactérien était formé d’ADN.Cette découverte, que l’on peut considérer comme l’acte de naissance de la biologie moléculaire ne fut pas, sauf exception, accueillie avec enthousiasme. Certes le travail expérimental était d’une rigueur exemplaire et les préparations obtenues ne permettaient d’y déceler aucun contaminant. Mais il restait toujours possible que des impuretés non décelées du fait de la sensibilité des méthodes employées fussent responsables de l’activité biologique. À l’origine de ces réticences se trouvait la théorie régnant à l’époque selon laquelle les acides nucléiques étaient de petites molécules formées par la succession monotone de groupe de 4 bases ATGC-ATGC, structure qui ne pouvait pas rendre compte de la richesse de l’information génétique. En fait, cette structure était déduite de l’analyse de fragments d’ADN très dégradés du fait des conditions de leur préparation.Cependant, la notion selon laquelle c’était bien l’ADN qui était porteur de l’information génétique devait s’imposer, d’une part, à la suite des recherches ultérieures de Avery et de ses collaborateurs, d’autre part, lorsque Hershey et Chase (1952) démontrèrent que c’est l’ADN des bactériophages (virus des bactéries) qui est injecté lorsque se produit la contamination virale dans la cellule bactérienne parasitée. Enfin Boivin, Vendrely et Vendrely, s’adressant cette fois à des organismes pluricellulaires montraient (1948) que les cellules diploïdes (les cellules somatiques de l’organisme qui contiennent 2 lots de chromosomes) contiennent par cellule deux fois plus d’ADN que les cellules haploïdes (les gamètes) qui ne contiennent qu’un seul lot de chromosomes.Le problème était donc de savoir quelles relations précises existaient entre gènes, acides nucléiques et protéines.La double héliceIl restait beaucoup d’inconnues: au niveau de la définition précise du gène, de la structure des acides nucléiques, de la structure des protéines, des rapports entre acides nucléiques et protéines.Le deuxième facteur important a été la mise en œuvre de techniques et de méthodes nouvelles: chromatographie et électrophorèse sur supports divers, mise au point de méthodes d’extraction et de purification des macromolécules non dénaturantes, utilisation des diagrammes de rayons X pour en déterminer la structure tridimensionnelle précise, microscopie électronique, techniques d’ultracentrifugation, etc.Ce sont ces méthodes qui permirent en particulier:– à Chargaff (1950) de déterminer avec précision la composition de molécules non dégradées d’ADN et d’en tirer les égalités: A = T; G = C: la teneur en adénine est égale à la teneur en thyamine; la teneur en guanine est égale à la teneur en cytosine.– à Sanger (1951-1955) de déterminer la séquence des acides aminés d’une protéine, l’insuline, révélant ainsi pour la première fois l’agencement spécifique des acides aminés d’une protéine donnée.– à Pauling dans les années cinquante de mettre en évidence les structures tridimensionnelles qu’imposent la géométrie des acides aminés et les liaisons de faible énergie (liaison hydrogène) qui maintiennent l’architecture des protéines et plus généralement des macromolécules.– à Perutz et Kendrew, après de nombreuses années de recherches, de définir à l’échelle de l’angström la structure tridimensionnelle de protéines comme la myoglobine puis l’hémoglobine déterminant ainsi (1961-1964) la position exacte de chacun des atomes de molécules qui en comportent plusieurs dizaines de milliers.C’est dans ce cadre qu’il convient de situer la découverte fondamentale par Watson et Crick de la structure des molécules d’ADN. À l’origine de l’étude cristallographique des protéines, inventeur du terme «biologie moléculaire», c’est W. T. Astbury qui fut le premier à s’intéresser à la structure tridimensionnelle de l’ADN. La détermination de la densité d’échantillons séchés d’ADN lui permit de conclure qu’il s’agissait d’une molécule filiforme et que les distances séparant les bases devaient être très petites: la molécule serait semblable à une colonne formée par l’empilement de bases les unes par-dessus les autres et dont les plans seraient perpendiculaires au grand axe de la molécule. Effectivement, et en dépit de la mauvaise qualité des diagrammes de rayons X obtenus à cette époque, Astbury put confirmer cette façon de voir et mesurer la distance entre les plateaux formés par les bases: 0,34 nm. Trois groupes de chercheurs prirent, au début des années 50, la relève de Astbury. Pauling que ses succès obtenus dans l’étude de la structure tridimensionnelle des protéines encourageaient à entreprendre celle de l’ADN ne put aboutir: la structure de l’ADN qu’il publia au début de l’année 1953 se révéla fausse. Un autre groupe était celui de Wilkins et son rôle fut déterminant car il parvint à préparer des fibres d’ADN remarquablement bien orientées. Entre les mains de Rosalind Franklin, ces préparations fournirent des photographies riches de détails qui, pour l’œil exercé d’un cristallographe se révélaient pleines d’enseignement, confirmant en particulier que la distance entre les plateaux de bases était bien de 0,34 nm. Watson et Crick qui avaient déjà envisagé diverses structures mais n’avaient pu conclure en raison de la mauvaise qualité des diagrammes dont ils disposaient, purent étudier les photographies de R. Franklin. En quelques semaines, ils mettaient au point leur modèle qui fut publié en avril 1953, dans Nature , en même temps que l’étaient les données cristallographiques de Wilkins, Stokes et Wilson (cf. fig. 7).Watson et Crick conclurent directement d’après les photographies que l’ADN avait une forme hélicoïdale, la molécule ayant un diamètre de 2 nm. D’autres données physico-chimiques (densité, courbe de titration...) les conduisirent à proposer que l’hélice était formée par la réunion de deux chaînes hélicoïdales dont le squelette était formé par les molécules de désoxyribose liées entre elles par les groupements phosphoriques: les plateaux de bases perpendiculaires au grand axe devaient donc se trouver à l’intérieur de la molécule.Une autre contrainte devait être prise en compte: après tout l’ADN est le matériel dont les gènes sont faits. On doit donc avoir affaire à un système tel qu’il s’accommode d’une très grande diversité. L’unique source de cette diversité ne peut être que l’ordre dans lequel les bases sont disposées les unes par rapport aux autres.Le problème était donc de construire une double hélice régulière qui, bien que constituée de deux chaînes où toute séquence de bases pouvait être représentée, aurait sur toute sa longueur un diamètre constant de 2 nm. Du fait que les dimensions des purines (adénine et guanine) sont plus grandes que celles des pyrimidines (cytosine et thymine), Watson et Crick eurent l’idée que la double hélice ne pouvait avoir ces propriétés que s’il existait une complémentarité telle qu’à tout niveau, si une des chaînes contient une purine, l’autre contient une pyrimidine. Selon quelles règles? Celles qui assurent le maximum de stabilité à l’édifice, c’est-à-dire la mise en place du plus grand nombre possible de liaisons hydrogène. Cela ne peut être réalisé (c’est l’examen des propriétés des molécules qui le montre) que si les bases complémentaires sont, d’une part, adénine-thymine, d’autre part, guanine-cytosine. C’est de cette façon, et seulement de cette façon, que, tout en maintenant constant le diamètre de la double hélice, on a le maximum de stabilité (deux liaisons hydrogène entre A et T, 3 entre G et C). Ainsi retrouve-t-on les égalités A = T, G = C mises en évidence par Chargaff et dont la signification était jusque-là demeurée assez mystérieuse. Mais l’importance capitale de cette découverte pour la compréhension des caractères essentiels des êtres vivants tient au fait que chaque molécule d’ADN est formée de deux chaînes qui sont complémentaires de par la séquence des bases qui les constituent. Chaque chaîne pouvant en effet servir de modèle, de matrice, pour la synthèse d’une chaîne complémentaire et donc identique à l’autre, on trouve là l’explication de la transmission aux générations successives de la même information génétique; si le message héréditaire est inscrit dans la séquence des bases, comme on le verra plus bas, les modifications de l’une d’entre elles pourront être à l’origine d’une mutation qui sera transmise à la descendance. C’est enfin, et encore, le même système d’appariements entre bases complémentaires A-T (ou A-U lorsqu’il s’agit d’acides ribonucléiques) et G-C qui gouverne le mode d’expression des gènes aboutissant, pour les gènes de structure spécifiant la synthèse de protéines, au positionnement dans un ordre défini de leurs acides aminés et, par voie de conséquence, aux structures tridimensionnelles d’où découlent les fonctions. Watson et Crick concluaient leur première publication par ces termes: «It has not escaped our notice that the specific pairing we have postulated immediately suggest a possible copying mechanism for the genetic material.» On appréciera la litote! Effectivement, dans les années qui suivirent, de nombreuses données expérimentales vinrent confirmer la justesse du modèle proposé que confirment encore journellement les travaux réalisés dans le domaine de la biologie moléculaire.Le modèle «Escherichia coli» + bactériophageLe troisième facteur important dans le développement de cette discipline a été le fait qu’à la suite de Luria et Delbrück, nombre de biologistes se sont intéressés à l’étude d’objets relativement simples, la bactérie Escherichia coli et ses bactériophages.Cette concentration des efforts sur un même matériel a été pour beaucoup dans les progrès réalisés. Il devenait possible de faire, à la fois, de la biochimie et de la génétique sur des organismes ou des virus se multipliant très rapidement et fournissant des populations homogènes de grande taille.C’est ce matériel qui permit, en particulier, à Benzer entre 1955 et 1961 d’expliciter la notion de gène, montrant que les trois définitions opérationnelles du gène comme unité de fonction, unité de recombinaison et unité de mutation ne désignent plus le même élément à un certain degré de finesse de l’analyse génétique: le gène redéfini comme unité de fonction (ou cistron) c’est-à-dire le plus petit élément qui doit rester intact, non muté, pour assurer sa fonction correspondant à une séquence longue d’environ 1 000 bases, la modification d’une seule de ces bases pouvant être à l’origine d’une mutation et des recombinaisons pouvant s’effectuer entre deux bases adjacentes. Ce qui fut confirmé en particulier par les recherches poursuivies par Yanofsky à partir de 1958 sur le contrôle génétique d’une enzyme intervenant dans la voie de biosynthèse du tryptophane: la tryptophane-synthétase. Cet auteur montrait, en outre, qu’une mutation peut avoir pour effet de conduire au remplacement d’un seul acide aminé par un autre, rendant la protéine non fonctionnelle. Il confirmait et généralisait ainsi les conclusions tirées par Ingram (1956-1960) de ses travaux sur les hémoglobines humaines des sujets atteints de cette forme d’anémie grave, héréditaire et sous le contrôle d’un seul gène, qu’est l’anémie à cellules falciformes (les globules rouges sont en forme de faucilles): dans l’hémoglobine normale, la chaîne 廓 de la globine porte en position 6 l’acide aminé acide glutamique; cet acide aminé (et c’est la seule différence) est remplacé par la valine dans le cas de l’anémie falciforme. Lorsque le dictionnaire des codons fut connu, il devint clair qu’il suffisait qu’une seule des trois bases spécifiant la mise en place d’un acide aminé soit modifiée pour qu’un autre acide aminé le remplace.La génétique d’E. coli et de ses phages fut élaborée à partir de la fin des années quarante par des auteurs tels que Lederberg, Hayes, Jacob et Wollman, et c’est grâce à ce matériel que Jacob et Monod (1961) étudièrent les phénomènes de régulation dans le fonctionnement des gènes, mettant en évidence l’existence de gènes opérateurs, d’opérons, de gènes régulateurs, d’ARN messagers intermédiaires entre l’ADN et les protéines, d’ARN messagers qui furent décelés et caractérisés, la même année, en particulier, d’une part, par F. Gros, d’autre part, par S. Spiegelman. Le fait de concentrer les efforts sur un même matériel a donc été pour beaucoup dans l’approfondissement des connaissances, et E. coli est toujours l’objet favori de nombreux chercheurs et l’outil indispensable des spécialistes du génie génétique.On notera cependant que d’autres matériels ont, pendant cette époque, et encore par la suite, fourni des résultats très intéressants. Par exemple, le virus de la mosaïque du tabac formé d’une molécule d’ARN englobée dans une coque protéique. Les deux composants peuvent être isolés l’un de l’autre: seul l’ARN est infectieux. Il est par ailleurs possible de faire se réassocier in vitro l’ARN provenant d’un virus avec les protéines d’un autre qui produit des symptômes différents: les symptômes provoqués par le virus reconstitué sont ceux du virus d’où provient l’ARN. Là encore des mutations ont pour effet de conduire au remplacement d’un seul acide aminé par un autre dans la protéine virale et, là encore, c’est le remplacement d’une seule des bases du codon concerné qui est mutagène (Fraenkel-Conrat Wittmann 1957-1961).La notion de codeUn dernier point concerne la notion de code. C’est semble-t-il E. Schrödinger qui, dans son très remarquable ouvrage intitulé What Is Life? (1944), l’a formulée: «It is these chromosomes, or probably only an axial skeleton fibre of what we actually see under the microscope as the chromosome, that contain in some kind of code-script the entire pattern of the individual’s future development and of its functioning in the mature state [...]. The chromosome structures are at the same time instrumental in bringing about the development they foreshadow. They are law-code and executive power-or, to use another simile, they are architect’s plan and builder’s craft – in one.» Après la publication de Watson et Crick, cette notion fut reprise de façon plus précise par l’astrophysicien Gamow qui l’exprima en ces termes: le patrimoine héréditaire de tout organisme sera assimilé à un mot (très long), écrit à l’aide d’un alphabet de 4 lettres. Les protéines seront considérées comme des mots écrits à l’aide d’un alphabet de 20 lettres.Posé ainsi en termes de décryptage, le problème allait faire l’objet de nombreuses publications théoriques, en particulier par Crick qui postulait l’intervention d’intermédiaires, d’adaptateurs, entre l’ARN messager et la protéine, adaptateurs qui devaient se révéler être les ARN de transfert.C’est finalement l’expérience qui permit de choisir entre toutes les solutions proposées, Crick montrant (1962) en n’utilisant que les méthodes génétiques que le message nucléique devait être lu, à partir d’un point de départ, toujours dans la même direction, par groupes de trois bases.Restaient à identifier chacun des groupes de trois bases, chacun des codons, responsables de la mise en place des acides aminés, c’est-à-dire établir le dictionnaire des codons.Les premières données furent obtenues par Nirenberg et Mattaei (1961). Quelques années auparavant M. Grunberg-Manago et Ochoa avaient mis en évidence une enzyme catalysant la synthèse in vitro de polymères ribonucléiques formés par la réunion d’une base UUUUU (acide polyuridylique (poly U); AAAA... (poly A) ou de plusieurs bases. Nirenberg et Mattaei travaillant sur des extraits de E. coli , dans un système contenant des ribosomes, une préparation acellulaire débarrassée d’ARN de haut poids moléculaire, des acides aminés, montrèrent que l’addition de poly U à ce système permet d’obtenir la synthèse d’une protéine artificielle uniquement formée de résidus phénylalanine: l’acide polyuridylique joue le rôle de messager artificiel et le codon correspondant à la phénylalanine doit être UUU, s’il s’agit bien de triplets. Une très intense activité s’ensuivit qui devait aboutir en très peu de temps à établir le dictionnaire des codons, la dernière touche étant apportée par Nirenberg et par Khorana (1965). Ce dernier prépara des ARN messagers de type GUGUGUGUGUG; la «protéine» synthétisée in vitro sous la direction de cet ARN est formée par l’alternance régulière de cystéine et de valine: cys - val - cys - val - ...: l’un des codons pour la cystéine est donc UGU, l’un des codons pour la valine est donc GUG.Et la conclusion essentielle que l’on tire de l’ensemble des résultats est que c’est bien l’ordre dans lequel les bases se trouvent disposées les unes par rapport aux autres qui est à l’origine de l’ordre dans lequel les acides aminés sont mis en place.3. Le présent et l’avenirL’étude des mécanismes biochimiques qui assurent le fonctionnement des êtres vivants, en particulier de ceux qui interviennent dans les grandes étapes que sont la réplication de l’ADN, sa transcription, la traduction des ARN messagers..., révèle pour chacune d’elles une très grande complexité avec l’intervention de plusieurs enzymes et de facteurs protéiques et autres. Dans bien des cas, de nombreuses questions continuent à se poser.Ne prenons qu’un seul exemple, celui de la transcription. Elle est assurée par un enzyme, l’ARN polymérase, formé par la réunion de plusieurs chaînes protéiques dont l’une, le facteur 靖, est indispensable pour le démarrage de la réaction lorsque l’ADN est sous forme de double hélice. L’enzyme ne réagit pas directement avec la première base de la séquence à transcrire mais avec des séquences particulières situées en amont et qui elles-mêmes ne sont pas transcrites. Ces séquences sont de longueur variable et définissent deux sites: un site de «reconnaissance» pour l’enzyme et un site où cette dernière se lie fortement, le site promoteur, dont la séquence est du type:
la mise en place d’1 acide aminé donné parmi les 20 qui composent les protéines est sous la dépendance d’un groupe de 3 bases parmi les 4 qui composent l’ADN, la spécificité étant assurée par les liaisons qui s’établissent entre les bases complémentaires adénine (A) et thymine (T) ou uracile (U), et guanine (G) et cytosine (C).C’est à l’ensemble des travaux qui portent sur la structure, le mode d’expression et le mode de reproduction des gènes qu’est souvent réservé, dans un sens plus restreint, le terme de biologie moléculaire. L’essentiel en est exposé ci-dessous.1. Bases de la biologie moléculaireL’acide désoxyribonucléique est fait de l’assemblage de motifs nucléotidiques , constitués par l’union d’un sucre, le désoxyribose, et d’une base nucléique (A, T, G ou C, c’est-à-dire adénine, thymine, guanine ou cytosine). Ces nucléotides sont reliés par l’intermédiaire d’un chaînon phosphoryle (P dans la figure 1 a).La chaîne phosphonucléotidique ainsi définie représente l’un des deux brins dont la molécule d’ADN est constituée: autrement dit, celle-ci est bicaténaire , avec deux brins accolés longitudinalement par des liaisons de faible énergie établies entre A et T, d’une part, C et G, d’autre part (fig. 1b). La complémentarité réciproque des deux brins conditionne donc la stabilité de cette structure. Dans ce cas, la molécule bicaténaire s’enroule en double hélice (fig. 7). Le fait que chacun des brins soit complémentaire de l’autre est dû aux modalités de la réplication de l’ADN.La bipartition qui va s’opérer à partir de la cellule mère, pour donner naissance à deux cellules filles, est précédée par la construction de constituants cellulaires qui seront donc partagés entre les cellules nouvelles. Dans le cas de l’ADN, chaque brin va servir de matrice pour la synthèse d’une chaîne complémentaire, qui en est donc la réplique . Ainsi se forme une nouvelle molécule (fig. 1c) dans laquelle l’un des brins est hérité de la molécule affectée par la réplication: celle-ci est donc semi-conservatrice. C’est pourquoi les cellules filles reçoivent l’une et l’autre des molécules d’ADN absolument identiques (à cette réserve près que le brin «ancien» de chacune d’elles n’est pas le même). Lorsque deux cellules filles ont reçu identiquement le même matériel génétique, elles synthétisent les mêmes protéines, et c’est pourquoi elles ont les mêmes structures et accomplissent les mêmes réactions chimiques.Mais, au contraire, en cas de mutation (fig. 1d), une partie du message se trouvant modifiée, la protéine régie par la séquence en cause sera différente, ce qui aura pour conséquence d’imposer un caractère nouveau à l’organisme et à ses descendants, à moins qu’une mutation «reverse» ne rétablisse l’ancien état des choses.Pour expliquer que la biosynthèse des protéines soit ainsi spécifiquement conjuguée à celles des acides nucléiques, il faut partir du fait que toute protéine est formée de chaînes d’acides aminés. Ceux-ci diffèrent par un radical R. Il en existe vingt espèces chimiques différentes selon les particularités dudit radical (R1...R20). La protéine renferme donc une séquence plus ou moins longue (une centaine à plusieurs centaines) d’acides aminés.Une fois synthétisée, ou au cours de sa synthèse, chacune de ces longues chaînes se replie sur elle-même, la structure tridimensionnelle adoptée ne dépendant que de la géométrie des atomes et étant stabilisée grâce aux liaisons hydrogènes: cette structure tridimensionnelle impose à la protéine sa fonction.Or l’ordre et la nature des acides aminés mis en place est sous la dépendance de l’ordre et de la nature des bases de l’ADN. Tout se passe comme s’il fallait traduire un alphabet à 4 lettres en un alphabet à 20 lettres afin que la coïncidence des deux séquences, nucléique et protéique, soit possible. L’expression génétique comprend ainsi deux étapes (fig. 2).Il y a d’abord une étape de transcription au cours de laquelle l’un des brins de l’ADN (tantôt l’un, tantôt l’autre, mais jamais les deux ensemble) sert de matrice pour la synthèse d’une chaîne complémentaire d’acide ribonucléique. Seules différences avec l’ADN: le sucre est ici le ribose et la thymine est remplacée par l’uracile. L’acide ribonucléique destiné à la biosynthèse des protéines est dit ARN messager (ARNm).Puis, une étape de traduction fait intervenir de nombreuses enzymes et de nombreux facteurs:– les ARN de transfert qui peuvent servir de «traducteurs» du fait qu’ils «connaissent» les deux langages, le langage nucléique et le langage protéique; un ARN de transfert donné peut en effet se lier de façon spécifique à un acide aminé donné (réaction catalysée par une enzyme spécifique); ce même ARN de transfert possède un groupe de trois bases (anti-codon) qui sont complémentaires d’un groupe de trois bases (codon) de l’ARN messager (fig. 3);– Les ribosomes, particules de nature ribonucléoprotéique, où se positionnent l’ARN messager et les ARN de transfert liés à leurs acides aminés, ce qui permet les liaisons entre ces acides aminés, les ARN de transfert étant détachés du complexe au fur et à mesure que chaque liaison s’effectue.La traduction s’effectue donc séquentiellement, à partir d’un point de départ et toujours dans la même direction, chaque acide aminé étant mis en place selon l’ordre qui lui est dicté par les séquences de codons de l’ARN messager qui dérive directement de la séquence des bases du segment de l’ADN du gène impliqué.La conception des mécanismes d’expression des gènes, qui vient d’être exposée, est issue de l’étude des bactéries et des virus, notamment les bactériophages. Les grandes étapes de ce processus, transcription et traduction, ont été retrouvées chez les organismes qui possèdent un véritable noyau, distinct du cytoplasme (les organismes eucaryotes) mais les modalités selon lesquelles ces processus s’y effectuent sont différentes, particulièrement chez les organismes pluricellulaires.Les différences portent sur plusieurs points, dont les principaux sont les suivants:– au contraire de ce qui se passe chez les bactéries, où la traduction du message (fig. 4) s’effectue dès qu’il commence à être synthétisé, dans les cellules eucaryotes le produit de transcription n’est pas immédiatement traduit, ce qui implique une certaine stabilité des ARN messagers;– le produit primaire de la transcription, qui se trouve dans le noyau cellulaire, est souvent (mais pas toujours) une molécule d’ARN géante comportant des séquences de bases qui ne seront pas traduites et qui est profondément remaniée avant d’aboutir à l’ARN messager proprement dit, selon le schéma de la figure 5.L’étude de molécules d’ARN intermédiaires entre le produit initial de la transcription de ces gènes dits «éclatés» ou «en mosaïque» et l’ARN messager, et qui permettent de saisir les étapes de la maturation, révèle que excisions et liaisons ne se font pas obligatoirement dans l’ordre où se situent les différentes séquences.Des séquences de bases (introns) n’auront pas codé pour l’ARN messager, elles n’en ont pas moins un rôle important. Il semble, en effet, que certaines d’entre elles soient nécessaires aux processus qui aboutissent à la molécule finale. C’est ainsi qu’une mutation intéressant l’un des introns du gène éclaté qui spécifie la synthèse de la chaîne 廓 de la globine humaine (et participe à l’édification de l’hémoglobine) provoque la formation d’ARN messager dont les propriétés ne sont pas normales. Le défaut dans la biosynthèse de la chaîne 廓 qui s’ensuit, entraîne une forme héréditaire de 廓 thalassémie, anémie grave.Il doit donc s’exercer au niveau des processus de maturation des ARN messagers des régulations dont les mécanismes sont actuellement à l’étude. Quoi qu’il en soit, les réactions mises en jeu dans l’excision des introns et l’aboutage des exons, sont d’une extrême précision. Un exemple en est fourni par le gène de l’insuline humaine (fig. 6).2. HistoriqueOn tentera ici, sans vouloir être exhaustif, de dégager les raisons des progrès très rapides qu’a connus et que connaît la biologie moléculaire en s’attachant plus particulièrement à ce qui touche l’étude de la structure et du mode de fonctionnement des gènes.L’hypothèse un gène (= acide nucléique) = une enzyme (= protéine)Tout d’abord, le problème était clairement posé. Dans les années cinquante, on savait déjà grâce aux travaux d’Ephrussi et Beadle (1935-1940) sur la Drosophile, de Beadle et Tatum (1940-1945) sur le champignon Neurospora que chaque réaction biochimique est sous le contrôle d’un gène donné et que ce contrôle s’exerce sur la protéine enzymatique qui catalyse la réaction. Ainsi se trouvaient confirmées les idées émises par Cuénot, d’une part, par Garrod, d’autre part, dès les premières années du siècle.Par ailleurs, Avery, Mac Leod et Mc Carty montraient en 1944 que le facteur mis en évidence par Griffith (1928) qui, extrait de pneumocoques virulents, confère de façon héréditaire cette virulence à des pneumocoques non virulents, est de l’ADN. Tout se passant comme si le ou les gènes gouvernant la biosynthèse de la capsule polysaccharidique des pneumocoques virulents avaient été transférés aux pneumocoques dépourvus de toute capsule et s’y exprimaient, ce qui signifiait que le gène bactérien était formé d’ADN.Cette découverte, que l’on peut considérer comme l’acte de naissance de la biologie moléculaire ne fut pas, sauf exception, accueillie avec enthousiasme. Certes le travail expérimental était d’une rigueur exemplaire et les préparations obtenues ne permettaient d’y déceler aucun contaminant. Mais il restait toujours possible que des impuretés non décelées du fait de la sensibilité des méthodes employées fussent responsables de l’activité biologique. À l’origine de ces réticences se trouvait la théorie régnant à l’époque selon laquelle les acides nucléiques étaient de petites molécules formées par la succession monotone de groupe de 4 bases ATGC-ATGC, structure qui ne pouvait pas rendre compte de la richesse de l’information génétique. En fait, cette structure était déduite de l’analyse de fragments d’ADN très dégradés du fait des conditions de leur préparation.Cependant, la notion selon laquelle c’était bien l’ADN qui était porteur de l’information génétique devait s’imposer, d’une part, à la suite des recherches ultérieures de Avery et de ses collaborateurs, d’autre part, lorsque Hershey et Chase (1952) démontrèrent que c’est l’ADN des bactériophages (virus des bactéries) qui est injecté lorsque se produit la contamination virale dans la cellule bactérienne parasitée. Enfin Boivin, Vendrely et Vendrely, s’adressant cette fois à des organismes pluricellulaires montraient (1948) que les cellules diploïdes (les cellules somatiques de l’organisme qui contiennent 2 lots de chromosomes) contiennent par cellule deux fois plus d’ADN que les cellules haploïdes (les gamètes) qui ne contiennent qu’un seul lot de chromosomes.Le problème était donc de savoir quelles relations précises existaient entre gènes, acides nucléiques et protéines.La double héliceIl restait beaucoup d’inconnues: au niveau de la définition précise du gène, de la structure des acides nucléiques, de la structure des protéines, des rapports entre acides nucléiques et protéines.Le deuxième facteur important a été la mise en œuvre de techniques et de méthodes nouvelles: chromatographie et électrophorèse sur supports divers, mise au point de méthodes d’extraction et de purification des macromolécules non dénaturantes, utilisation des diagrammes de rayons X pour en déterminer la structure tridimensionnelle précise, microscopie électronique, techniques d’ultracentrifugation, etc.Ce sont ces méthodes qui permirent en particulier:– à Chargaff (1950) de déterminer avec précision la composition de molécules non dégradées d’ADN et d’en tirer les égalités: A = T; G = C: la teneur en adénine est égale à la teneur en thyamine; la teneur en guanine est égale à la teneur en cytosine.– à Sanger (1951-1955) de déterminer la séquence des acides aminés d’une protéine, l’insuline, révélant ainsi pour la première fois l’agencement spécifique des acides aminés d’une protéine donnée.– à Pauling dans les années cinquante de mettre en évidence les structures tridimensionnelles qu’imposent la géométrie des acides aminés et les liaisons de faible énergie (liaison hydrogène) qui maintiennent l’architecture des protéines et plus généralement des macromolécules.– à Perutz et Kendrew, après de nombreuses années de recherches, de définir à l’échelle de l’angström la structure tridimensionnelle de protéines comme la myoglobine puis l’hémoglobine déterminant ainsi (1961-1964) la position exacte de chacun des atomes de molécules qui en comportent plusieurs dizaines de milliers.C’est dans ce cadre qu’il convient de situer la découverte fondamentale par Watson et Crick de la structure des molécules d’ADN. À l’origine de l’étude cristallographique des protéines, inventeur du terme «biologie moléculaire», c’est W. T. Astbury qui fut le premier à s’intéresser à la structure tridimensionnelle de l’ADN. La détermination de la densité d’échantillons séchés d’ADN lui permit de conclure qu’il s’agissait d’une molécule filiforme et que les distances séparant les bases devaient être très petites: la molécule serait semblable à une colonne formée par l’empilement de bases les unes par-dessus les autres et dont les plans seraient perpendiculaires au grand axe de la molécule. Effectivement, et en dépit de la mauvaise qualité des diagrammes de rayons X obtenus à cette époque, Astbury put confirmer cette façon de voir et mesurer la distance entre les plateaux formés par les bases: 0,34 nm. Trois groupes de chercheurs prirent, au début des années 50, la relève de Astbury. Pauling que ses succès obtenus dans l’étude de la structure tridimensionnelle des protéines encourageaient à entreprendre celle de l’ADN ne put aboutir: la structure de l’ADN qu’il publia au début de l’année 1953 se révéla fausse. Un autre groupe était celui de Wilkins et son rôle fut déterminant car il parvint à préparer des fibres d’ADN remarquablement bien orientées. Entre les mains de Rosalind Franklin, ces préparations fournirent des photographies riches de détails qui, pour l’œil exercé d’un cristallographe se révélaient pleines d’enseignement, confirmant en particulier que la distance entre les plateaux de bases était bien de 0,34 nm. Watson et Crick qui avaient déjà envisagé diverses structures mais n’avaient pu conclure en raison de la mauvaise qualité des diagrammes dont ils disposaient, purent étudier les photographies de R. Franklin. En quelques semaines, ils mettaient au point leur modèle qui fut publié en avril 1953, dans Nature , en même temps que l’étaient les données cristallographiques de Wilkins, Stokes et Wilson (cf. fig. 7).Watson et Crick conclurent directement d’après les photographies que l’ADN avait une forme hélicoïdale, la molécule ayant un diamètre de 2 nm. D’autres données physico-chimiques (densité, courbe de titration...) les conduisirent à proposer que l’hélice était formée par la réunion de deux chaînes hélicoïdales dont le squelette était formé par les molécules de désoxyribose liées entre elles par les groupements phosphoriques: les plateaux de bases perpendiculaires au grand axe devaient donc se trouver à l’intérieur de la molécule.Une autre contrainte devait être prise en compte: après tout l’ADN est le matériel dont les gènes sont faits. On doit donc avoir affaire à un système tel qu’il s’accommode d’une très grande diversité. L’unique source de cette diversité ne peut être que l’ordre dans lequel les bases sont disposées les unes par rapport aux autres.Le problème était donc de construire une double hélice régulière qui, bien que constituée de deux chaînes où toute séquence de bases pouvait être représentée, aurait sur toute sa longueur un diamètre constant de 2 nm. Du fait que les dimensions des purines (adénine et guanine) sont plus grandes que celles des pyrimidines (cytosine et thymine), Watson et Crick eurent l’idée que la double hélice ne pouvait avoir ces propriétés que s’il existait une complémentarité telle qu’à tout niveau, si une des chaînes contient une purine, l’autre contient une pyrimidine. Selon quelles règles? Celles qui assurent le maximum de stabilité à l’édifice, c’est-à-dire la mise en place du plus grand nombre possible de liaisons hydrogène. Cela ne peut être réalisé (c’est l’examen des propriétés des molécules qui le montre) que si les bases complémentaires sont, d’une part, adénine-thymine, d’autre part, guanine-cytosine. C’est de cette façon, et seulement de cette façon, que, tout en maintenant constant le diamètre de la double hélice, on a le maximum de stabilité (deux liaisons hydrogène entre A et T, 3 entre G et C). Ainsi retrouve-t-on les égalités A = T, G = C mises en évidence par Chargaff et dont la signification était jusque-là demeurée assez mystérieuse. Mais l’importance capitale de cette découverte pour la compréhension des caractères essentiels des êtres vivants tient au fait que chaque molécule d’ADN est formée de deux chaînes qui sont complémentaires de par la séquence des bases qui les constituent. Chaque chaîne pouvant en effet servir de modèle, de matrice, pour la synthèse d’une chaîne complémentaire et donc identique à l’autre, on trouve là l’explication de la transmission aux générations successives de la même information génétique; si le message héréditaire est inscrit dans la séquence des bases, comme on le verra plus bas, les modifications de l’une d’entre elles pourront être à l’origine d’une mutation qui sera transmise à la descendance. C’est enfin, et encore, le même système d’appariements entre bases complémentaires A-T (ou A-U lorsqu’il s’agit d’acides ribonucléiques) et G-C qui gouverne le mode d’expression des gènes aboutissant, pour les gènes de structure spécifiant la synthèse de protéines, au positionnement dans un ordre défini de leurs acides aminés et, par voie de conséquence, aux structures tridimensionnelles d’où découlent les fonctions. Watson et Crick concluaient leur première publication par ces termes: «It has not escaped our notice that the specific pairing we have postulated immediately suggest a possible copying mechanism for the genetic material.» On appréciera la litote! Effectivement, dans les années qui suivirent, de nombreuses données expérimentales vinrent confirmer la justesse du modèle proposé que confirment encore journellement les travaux réalisés dans le domaine de la biologie moléculaire.Le modèle «Escherichia coli» + bactériophageLe troisième facteur important dans le développement de cette discipline a été le fait qu’à la suite de Luria et Delbrück, nombre de biologistes se sont intéressés à l’étude d’objets relativement simples, la bactérie Escherichia coli et ses bactériophages.Cette concentration des efforts sur un même matériel a été pour beaucoup dans les progrès réalisés. Il devenait possible de faire, à la fois, de la biochimie et de la génétique sur des organismes ou des virus se multipliant très rapidement et fournissant des populations homogènes de grande taille.C’est ce matériel qui permit, en particulier, à Benzer entre 1955 et 1961 d’expliciter la notion de gène, montrant que les trois définitions opérationnelles du gène comme unité de fonction, unité de recombinaison et unité de mutation ne désignent plus le même élément à un certain degré de finesse de l’analyse génétique: le gène redéfini comme unité de fonction (ou cistron) c’est-à-dire le plus petit élément qui doit rester intact, non muté, pour assurer sa fonction correspondant à une séquence longue d’environ 1 000 bases, la modification d’une seule de ces bases pouvant être à l’origine d’une mutation et des recombinaisons pouvant s’effectuer entre deux bases adjacentes. Ce qui fut confirmé en particulier par les recherches poursuivies par Yanofsky à partir de 1958 sur le contrôle génétique d’une enzyme intervenant dans la voie de biosynthèse du tryptophane: la tryptophane-synthétase. Cet auteur montrait, en outre, qu’une mutation peut avoir pour effet de conduire au remplacement d’un seul acide aminé par un autre, rendant la protéine non fonctionnelle. Il confirmait et généralisait ainsi les conclusions tirées par Ingram (1956-1960) de ses travaux sur les hémoglobines humaines des sujets atteints de cette forme d’anémie grave, héréditaire et sous le contrôle d’un seul gène, qu’est l’anémie à cellules falciformes (les globules rouges sont en forme de faucilles): dans l’hémoglobine normale, la chaîne 廓 de la globine porte en position 6 l’acide aminé acide glutamique; cet acide aminé (et c’est la seule différence) est remplacé par la valine dans le cas de l’anémie falciforme. Lorsque le dictionnaire des codons fut connu, il devint clair qu’il suffisait qu’une seule des trois bases spécifiant la mise en place d’un acide aminé soit modifiée pour qu’un autre acide aminé le remplace.La génétique d’E. coli et de ses phages fut élaborée à partir de la fin des années quarante par des auteurs tels que Lederberg, Hayes, Jacob et Wollman, et c’est grâce à ce matériel que Jacob et Monod (1961) étudièrent les phénomènes de régulation dans le fonctionnement des gènes, mettant en évidence l’existence de gènes opérateurs, d’opérons, de gènes régulateurs, d’ARN messagers intermédiaires entre l’ADN et les protéines, d’ARN messagers qui furent décelés et caractérisés, la même année, en particulier, d’une part, par F. Gros, d’autre part, par S. Spiegelman. Le fait de concentrer les efforts sur un même matériel a donc été pour beaucoup dans l’approfondissement des connaissances, et E. coli est toujours l’objet favori de nombreux chercheurs et l’outil indispensable des spécialistes du génie génétique.On notera cependant que d’autres matériels ont, pendant cette époque, et encore par la suite, fourni des résultats très intéressants. Par exemple, le virus de la mosaïque du tabac formé d’une molécule d’ARN englobée dans une coque protéique. Les deux composants peuvent être isolés l’un de l’autre: seul l’ARN est infectieux. Il est par ailleurs possible de faire se réassocier in vitro l’ARN provenant d’un virus avec les protéines d’un autre qui produit des symptômes différents: les symptômes provoqués par le virus reconstitué sont ceux du virus d’où provient l’ARN. Là encore des mutations ont pour effet de conduire au remplacement d’un seul acide aminé par un autre dans la protéine virale et, là encore, c’est le remplacement d’une seule des bases du codon concerné qui est mutagène (Fraenkel-Conrat Wittmann 1957-1961).La notion de codeUn dernier point concerne la notion de code. C’est semble-t-il E. Schrödinger qui, dans son très remarquable ouvrage intitulé What Is Life? (1944), l’a formulée: «It is these chromosomes, or probably only an axial skeleton fibre of what we actually see under the microscope as the chromosome, that contain in some kind of code-script the entire pattern of the individual’s future development and of its functioning in the mature state [...]. The chromosome structures are at the same time instrumental in bringing about the development they foreshadow. They are law-code and executive power-or, to use another simile, they are architect’s plan and builder’s craft – in one.» Après la publication de Watson et Crick, cette notion fut reprise de façon plus précise par l’astrophysicien Gamow qui l’exprima en ces termes: le patrimoine héréditaire de tout organisme sera assimilé à un mot (très long), écrit à l’aide d’un alphabet de 4 lettres. Les protéines seront considérées comme des mots écrits à l’aide d’un alphabet de 20 lettres.Posé ainsi en termes de décryptage, le problème allait faire l’objet de nombreuses publications théoriques, en particulier par Crick qui postulait l’intervention d’intermédiaires, d’adaptateurs, entre l’ARN messager et la protéine, adaptateurs qui devaient se révéler être les ARN de transfert.C’est finalement l’expérience qui permit de choisir entre toutes les solutions proposées, Crick montrant (1962) en n’utilisant que les méthodes génétiques que le message nucléique devait être lu, à partir d’un point de départ, toujours dans la même direction, par groupes de trois bases.Restaient à identifier chacun des groupes de trois bases, chacun des codons, responsables de la mise en place des acides aminés, c’est-à-dire établir le dictionnaire des codons.Les premières données furent obtenues par Nirenberg et Mattaei (1961). Quelques années auparavant M. Grunberg-Manago et Ochoa avaient mis en évidence une enzyme catalysant la synthèse in vitro de polymères ribonucléiques formés par la réunion d’une base UUUUU (acide polyuridylique (poly U); AAAA... (poly A) ou de plusieurs bases. Nirenberg et Mattaei travaillant sur des extraits de E. coli , dans un système contenant des ribosomes, une préparation acellulaire débarrassée d’ARN de haut poids moléculaire, des acides aminés, montrèrent que l’addition de poly U à ce système permet d’obtenir la synthèse d’une protéine artificielle uniquement formée de résidus phénylalanine: l’acide polyuridylique joue le rôle de messager artificiel et le codon correspondant à la phénylalanine doit être UUU, s’il s’agit bien de triplets. Une très intense activité s’ensuivit qui devait aboutir en très peu de temps à établir le dictionnaire des codons, la dernière touche étant apportée par Nirenberg et par Khorana (1965). Ce dernier prépara des ARN messagers de type GUGUGUGUGUG; la «protéine» synthétisée in vitro sous la direction de cet ARN est formée par l’alternance régulière de cystéine et de valine: cys - val - cys - val - ...: l’un des codons pour la cystéine est donc UGU, l’un des codons pour la valine est donc GUG.Et la conclusion essentielle que l’on tire de l’ensemble des résultats est que c’est bien l’ordre dans lequel les bases se trouvent disposées les unes par rapport aux autres qui est à l’origine de l’ordre dans lequel les acides aminés sont mis en place.3. Le présent et l’avenirL’étude des mécanismes biochimiques qui assurent le fonctionnement des êtres vivants, en particulier de ceux qui interviennent dans les grandes étapes que sont la réplication de l’ADN, sa transcription, la traduction des ARN messagers..., révèle pour chacune d’elles une très grande complexité avec l’intervention de plusieurs enzymes et de facteurs protéiques et autres. Dans bien des cas, de nombreuses questions continuent à se poser.Ne prenons qu’un seul exemple, celui de la transcription. Elle est assurée par un enzyme, l’ARN polymérase, formé par la réunion de plusieurs chaînes protéiques dont l’une, le facteur 靖, est indispensable pour le démarrage de la réaction lorsque l’ADN est sous forme de double hélice. L’enzyme ne réagit pas directement avec la première base de la séquence à transcrire mais avec des séquences particulières situées en amont et qui elles-mêmes ne sont pas transcrites. Ces séquences sont de longueur variable et définissent deux sites: un site de «reconnaissance» pour l’enzyme et un site où cette dernière se lie fortement, le site promoteur, dont la séquence est du type: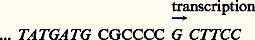 c’est au niveau de tels sites promoteurs que se fixent les répresseurs ou les activateurs de nature protéique identifiés chez les bactéries et qui commandent, en entrant ou non en compétition avec l’ARN polymérase le démarrage ou le non-démarrage de la synthèse de l’ARN concerné, autrement dit l’expression ou la mise au repos des gènes correspondants. On ne sait en revanche que peu de chose de la nature exacte des signaux nucléiques responsables de la fin d’une transcription, sinon que, là encore, il est vraisemblable qu’interviennent des protéines telles que celle qui a été isolée de E. coli : elle est nécessaire in vitro, dans un système de transcription reconstitué à la production de l’ARN de longueur normale tel qu’il est synthétisé in vivo; en son absence, les ARN produits sont anormalement longs.Élucider les mécanismes d’interactions entre séquences de bases nucléiques et protéines est d’une importance capitale pour la compréhension des phénomènes de régulation de l’expression des gènes. Car c’est ainsi que se pose le problème des mécanismes moléculaires qui fondent le déroulement des programmes de développement et de différenciation qui de l’œuf, ou de la graine, conduisent à la formation d’organismes pluricellulaires: certains gènes, ou groupes de gènes s’exprimant à certains stades précis du développement alors que d’autres sont mis «au repos».La structure fine de l’ADNCertaines méthodes physiques comme la diffraction des rayons X et des neutrons, l’utilisation du rayonnement synchrotron qui permet de réaliser des études tant dynamiques que structurales, les diverses techniques de la spectroscopie... fournissent déjà à cet égard des renseignements fort intéressants. Elles révèlent en particulier que ni les acides nucléiques ni les protéines ne sont des structures figées et que leurs interactions (ainsi qu’avec d’autres molécules) mettent en jeu des modifications de leur architecture, de leur conformation.Dans le même ordre d’idées, l’étude détaillée de la structure de la double hélice permet de montrer qu’elle n’est pas aussi régulière qu’on le pensait: elle présente de légères variations qui dépendent de la séquence des bases. Certaines de ces «irrégularités» sont reconnues par des protéines. On arrive ainsi à la notion qui se dégageait déjà de l’étude des protéines, selon laquelle la conformation de la molécule imposée par l’enchaînement de ses composants joue un rôle important pour ses propriétés biologiques. Cette notion se trouve confirmée par la mise en évidence d’enzymes qui modifient la topologie de la molécule, et par la mise en évidence de segments en hélice gauche alors que le modèle de Watson et Crick était en hélice droite, sur toute sa longueur. Enfin, il apparaît clairement que l’ADN est une structure dynamique sujette à des changements réversibles de conformation. Autant de données qui laissent prévoir des développements importants pour la connaissance des propriétés des macromolécules qui fondent leurs interactions.Identification des séquencesÀ partir des années soixante-dix, d’autres méthodes, de nouvelles techniques ont été élaborées qui ont fourni des résultats aussi intéressants que parfois inattendus. Citons parmi les principales:– La séparation des protéines par électrophorèse en double dimension donne la possibilité d’analyser qualitativement et quantitativement la plus grande partie sinon la totalité des protéines qui se trouvent ou qui sont synthétisées, par exemple, aux différents stades du développement d’un organisme donné; sa sensibilité est telle qu’elle peut révéler l’absence d’une seule protéine (parmi les milliers qu’elle contient) dans des cellules humaines mutantes ou des différences dans une seule protéine présente mais modifiée à la suite d’une mutation;– Les ARN messagers peuvent être isolés, purifiés, et leur traduction peut s’effectuer in vitro. Les protéines synthétisées dans ces conditions peuvent être comparées à celles qui le sont par des cellules intactes, ce qui fournit des renseignements sur les types de régulation qui opèrent au niveau de la traduction, au cours de la différenciation, par exemple;– L’hybridation moléculaire fondée sur la complémentarité des bases A-T (U); G-C. Elle fournit des renseignements précieux tant sur la structure des génomes quand il s’agit d’hybridation entre ADN et ADN, que sur les modalités de leur expression dans le cas des hybridations ADN-ARN. La méthode d’hybridation ADN-ADN a ainsi révélé qu’au contraire de ce qui se passe chez les bactéries, le génome des cellules eucaryotes est composite, formé de séquences semblables qui se trouvent répétées en de nombreux exemplaires et de séquences qui ne se trouvent représentées qu’une fois et dont font partie les gènes spécifiant la synthèse des protéines; cette même méthode permet d’isoler et de purifier les différentes classes de séquences; quant aux hybridations ADN-ARN, elles permettent, par exemple, de savoir avec quelle fréquence tel type de séquence d’ADN est transcrite, de déceler les espèces d’ARN spécifiques d’un état différencié donné, d’en mesurer les quantités synthétisées, de comparer entre eux les résultats obtenus sur différents types cellulaires depuis l’œuf, quand le matériel s’y prête, jusqu’aux états différenciés terminaux. C’est enfin l’hybridation moléculaire qui permet de repérer parmi les lignées bactériennes où se trouvent insérés les fragments d’ADN d’un autre organisme, celle qui porte le gène auquel on s’intéresse, le repérage se faisant par le biais d’une sonde radioactive (ARN ou ADN complémentaire, synthétisé à partir de ce dernier) qui s’hybride de façon sélective avec l’ADN recherché;– L’ADN peut être découpé en petits fragments grâce à des enzymes spécifiques, des endonucléases (ou enzymes de restriction) qui ont la propriété de scinder la molécule en des points précis de sa structure déterminés par la séquence de quelques nucléotides qui sont adjacents aux sites d’action des enzymes. Il est ainsi possible d’obtenir des fragments de petite taille que l’on peut isoler et caractériser, et d’établir une carte physique de la molécule par recoupement des fragments obtenus en faisant agir plusieurs nucléases qui diffèrent par la séquence des bases de leurs sites d’attachement. L’obtention de ces fragments courts rend possible l’analyse de la séquence des bases dont ils sont constitués. De nouvelles méthodes fiables et précises ont été élaborées qui permettent de déterminer la séquence exacte de plusieurs dizaines de milliers de bases.Isolement d’un gèneMais ces techniques n’auraient qu’un intérêt relativement limité s’il n’était pas possible d’avoir à l’état pur et en quantité suffisante le gène dont on veut étudier les propriétés. Or, chez un organisme pluricellulaire, tel que l’homme, par exemple, un gène donné ne représente que le millionième de l’ADN. D’où l’intérêt des techniques de clonage, base du génie génétique. Dans leur principe, elles consistent, après avoir fragmenté l’ADN, à insérer chacun des fragments dans l’ADN d’un vecteur (virus ou plasmide) capable de se répliquer dans une bactérie qui, en général, est E. coli . Le vecteur porte un gène qui confère la résistance des bactéries à un antibiotique. Le traitement de la population bactérienne par cet antibiotique aura pour effet de sélectionner celles qui sont résistantes du fait qu’elles hébergent bien l’ADN du vecteur lié au génome à étudier. Au pouvoir de multiplication du vecteur dans chaque cellule bactérienne, s’ajoute celui des bactéries elles-mêmes qui fournissent des populations aussi grandes qu’on le désire: le génome dont on veut étudier la structure et les propriétés se trouve donc amplifié de façon considérable. C’est ainsi que le génome humain, celui de la souris, de la drosophile... sont répartis dans différentes lignées, dans différents clones d’E. coli . Les fragmentations ayant été opérées de façon statistique (par voie enzymatique ou par voie mécanique), il y a une chance non négligeable pour que le gène auquel on s’intéresse soit en partie, en totalité (seul ou encore lié à ses segments adjacents), dans un ou quelques clones bactériens. Comment repérer ce ou ces clones? Soit par le produit de son expression... si le gène veut bien s’exprimer dans la bactérie soit, d’une façon plus générale, à l’aide d’une sonde. Celle-ci est constituée par l’ARN messager, rendu radioactif, qui spécifie la synthèse de la protéine qui est sous le contrôle du gène. Les techniques d’hybridation moléculaire permettent alors de repérer parmi tous les clones bactériens, le et seulement le clone qui contient le gène en question. Une variante de cette méthode consiste à utiliser comme sonde la molécule d’ADN radioactive synthétisée in vitro à partir de l’ARN messager pris comme matrice, à l’aide d’une enzyme, la transcriptase-réserve: on dispose ainsi d’une quantité importante de la molécule utilisée comme sonde, alors que l’ARN messager lui-même ne peut en général être obtenu qu’en petite quantité.Une fois le clone bactérien repéré, il suffira d’en extraire le vecteur et d’en libérer le gène en traitant ce vecteur par des enzymes de restriction.Les méthodes qui viennent d’être décrites, auxquelles il convient d’associer celles qui font appel à l’étude en microscopie électronique des molécules d’ADN, d’ARN et de leurs hybrides sont à l’origine d’une moisson de résultats dont certains exemples ont déjà été donnés (gènes éclatés, structure du gène de l’insuline (fig. 6), séquence des promoteurs...). Outre l’intérêt qu’elles présentent dans le domaine des biotechnologies (synthèse bactérienne d’insuline humaine ou d’autres hormones, de motifs antigéniques pour la production de nouveaux vaccins, étude des gènes qui contrôlent la fixation de l’azote chez les bactéries ou chez les symbiotes des plantes...), elles constituent un outil d’une finesse remarquable dans l’analyse des propriétés des êtres vivants.Contrôle des systèmes codantsOn en trouvera ici quelques exemples:D’abord, le fait de pouvoir isoler un gène avec ses parties adjacentes et d’en déterminer les séquences, permet d’accéder aux groupes de bases qui, situées à l’extérieur du gène proprement dit peuvent jouer le rôle de sites de reconnaissance pour les protéines régulatrices. Il est possible par voie chimique, de modifier directement et à volonté une base donnée, de faire par conséquent de la mutagenèse dirigée. L’étude des répercussions sur les propriétés fonctionnelles de la molécule permet de bien mieux les comprendre.Par ailleurs, connaissant la séquence des bases, il est possible d’en déduire la séquence des acides aminés de la protéine dont la synthèse est régie par le gène, méthode rapide et sûre quand il s’agit de protéines de poids moléculaire élevé dont l’analyse conventionnelle est difficile et qui fournit parfois des résultats intéressants en ce qu’elle révèle l’existence de séquences d’acides aminés qui seront éliminées lors de la transformation de la protéine synthétisée en protéine fonctionnelle. À l’inverse, connaissant la séquence de quelques acides aminés d’une protéine, et en déduisant la séquence des bases, on peut synthétiser par voie chimique la partie correspondante du gène. Celle-ci sera utilisée comme sonde: on évite ainsi l’isolement de l’ARN messager qui, sauf exception, est souvent une entreprise longue et délicate.Il est important, enfin, dans le cas des maladies héréditaires graves et invalidantes, et pour lesquelles aucun traitement n’est connu, de pouvoir prédire si l’enfant à naître présentera ou non cette maladie de façon, si c’est le cas, à pouvoir proposer un avortement aux parents. C’est le cas, par exemple, pour les anémies graves comme les thalassémies qui affectent la fonction de l’hémoglobine. Les gènes qui spécifient la synthèse des protéines constituants de cette molécule sont isolés. Utilisés comme sondes, ils ont permis de montrer que, dans certains cas, les troubles avaient pour origine la perte plus ou moins grande (parfois totale) du gène correspondant. L’étude d’ADN des cellules du fœtus, au moyen des enzymes de restriction permet de savoir si l’enfant à naître souffrira ou non de la maladie. La même méthodologie appliquée à l’anémie falciforme permet de tirer de semblables conclusions. Dans ce système des hémoglobines humaines, les études de génétique formelle et de génétique moléculaire aboutissent à dresser de façon précise la carte des gènes impliqués, montrant en particulier que les différents types d’hémoglobine apparaissent au cours du développement selon l’ordre dans lequel ces gènes sont situés les uns par rapport aux autres, et révélant l’existence de séquences en partie homologues qui ne sont pas exprimées.Instabilité du génomeL’une des découvertes les plus surprenantes concerne la relative instabilité du matériel génétique, aussi bien chez les procaryotes que chez les eucaryotes. Les modèles moléculaires d’ADN, ses photographies en microscopie électronique en donnent une image figée et statique qui ne correspond pas à la réalité. En fait, l’ADN est constamment soumis à des remaniements plus ou moins profonds: des gènes sont transposés d’un chromosome à un autre, des segments d’ADN sont insérés dans la molécule, d’autres en sont excisés, des recombinaisons s’effectuent, des lésions sont provoquées, qui sont réparées par des enzymes. On a donc affaire à un système qui se renouvelle. Quelques exemples permettront d’illustrer ce point.L’étude de trypanosomes, comme ceux qui sont à l’origine de la maladie du sommeil, avait montré que ces parasites échappent aux défenses immunitaires de l’hôte par le mécanisme suivant: lors de la première infection, l’hôte élabore des anticorps contre un déterminant antigénique du parasite. Celui-ci (ou une partie de la population) élabore alors rapidement un nouveau déterminant et le phénomène se reproduit. À ce jeu d’attaque et de réponse, l’hôte est perdant. D’où, en particulier, la difficulté de préparer des vaccins. Certaines des causes de cette diversité dans la production d’antigènes ont été élucidées lorsque les méthodes de la biologie moléculaire ont été appliquées à ce système. Le génome des trypanosomes contient une centaine d’exemplaires de séquences homologues mais qui diffèrent légèrement les unes des autres. Une seule de ces séquences, celle qui se trouve à proximité d’un site d’activation est exprimée à chaque fois, aboutissant à la synthèse d’un déterminant antigénique. En présence de l’anticorps formé contre ce déterminant, et par un mécanisme de transposition, la séquence qui s’exprimait est excisée et elle est remplacée par une copie de l’une de celles qui se trouvaient au repos et dont l’expression va se trouver déclenchée.Restant dans le domaine de l’immunologie, ce sont encore les méthodes de génie génétique qui ont permis de comprendre, au moins en grande partie, l’origine de l’extraordinaire diversité des anticorps. L’étude comparée de l’organisation des gènes concernés dans les cellules germinales et dans les cellules productrices d’anticorps révèle qu’au cours des processus de différenciation qui aboutissent à ces cellules compétentes (dont chacune n’exprime qu’un seul anticorps), de profonds remaniements interviennent. Ces remaniements se produisent au niveau de l’ADN, aboutissant à l’excision de séquences et à l’aboutage d’autres selon des combinaisons extrêmement diverses. À cela s’ajoutent, augmentant encore la diversité, des mutations somatiques et, au niveau, cette fois, des produits de transcription, de nouveaux remaniements qui sont caractéristiques des gènes en mosaïque. Un problème biologique fondamental se trouve ainsi éclairé d’un jour tout à fait nouveau.ÉvolutionUne des conséquences importantes de la mise en évidence de la relative instabilité de l’ADN concerne l’étude des mécanismes de l’évolution; un autre touche aux processus de différenciation.Pour évoluer, il est nécessaire de créer du nouveau, tout en restant viable et capable de transmettre la nouveauté à la descendance. Mutations de gènes et recombinaisons entre gènes étaient des mécanismes déjà connus des origines de la variabilité; de même que l’était le mode d’apparition de nouveaux gènes, copies formées à la suite d’accidents survenant lors de l’appariement des chromosomes homologues au cours de la méïose (crossing-over inégaux ou réparations à l’origine de la conversion, du retour d’une séquence mutée vers la séquence normale ou vice-versa). Les séquences des copies peuvent être modifiées tandis que les gènes d’origine continuent à assurer les fonctions habituelles de l’organisme. Si, à la suite de ces modifications, les nouveaux gènes confèrent, par exemple, un avantage aux organismes qui les portent, si elles se trouvent transposées à proximité de sites qui activent leur expression, elles pourront se répandre dans la population, être à l’origine de la manifestation de nouveaux caractères, de la mise en place de barrières génétiques entre les organismes parentaux et certains de leurs descendants, conduire à l’apparition de nouvelles espèces.Les arguments expérimentaux ne manquent pas, qui appuient ces conceptions et permettent d’aborder un aspect nouveau de la génétique des populations.Isoler un gène donné d’une espèce donnée, en déterminer la séquence, l’utiliser comme sonde chez les organismes appartenant à la même espèce, permet d’y déceler des séquences homologues légèrement modifiées, de mettre en évidence, par conséquent, un polymorphisme au niveau de l’ADN. Cela d’une façon plus directe et plus fiable que celle qui repose sur la comparaison de la séquence des acides aminés de protéines homologues et surtout en ayant accès à des séquences qui ne sont pas transcrites, comme les séquences régulatrices, ou qui, transcrites, ne sont pas traduites comme les introns. C’est ainsi qu’ont été mises en évidence des séquences proches de celles qui spécifient la synthèse des différentes globines qui entrent dans la composition des hémoglobines et qui, situées à proximité de celles-ci, ne s’expriment pas: les pseudo-gènes. C’est aussi ce type d’approche qui renforce la conception selon laquelle un gène ancestral unique serait à l’origine des différents gènes qui opèrent actuellement dans ce système.La même méthodologie permet de comparer les séquences de gènes homologues dans différentes espèces, de juger de leur conservation ou de leur vitesse d’évolution. Un exemple en a déjà été fourni, celui du gène de l’insuline humaine et d’un des gènes de l’insuline du rat: dans ce cas, les séquences ont peu évolué l’une par rapport à l’autre; en revanche l’autre gène de l’insuline du rat a évolué, lui, de façon indépendante et marquée. Dans le même ordre d’idées, les gènes des histones (protéines associées à l’ADN des cellules eucaryotes) et dont il existe de très nombreuses copies chez un même organisme, présentent un très haut degré d’homologie chez tous les organismes où ils existent, ce qui plaide en faveur de leur importance dans l’économie de la cellule. Le fait que ces séquences aient été conservées doit signifier que la structure des protéines spécifiées ne peut varier que dans de très étroites limites sous peine de voir leurs fonctions abolies.Différenciation - oncogenèseUn dernier exemple, et qui concerne tout autant les processus de différenciation, est celui des séquences oncogènes. Il s’agit de séquences de l’ADN cellulaire homologue aux gènes de virus qui ont la propriété de transformer des cellules normales en cellules cancéreuses. Ces gènes ont vu leurs séquences remarquablement bien conservées au cours de l’évolution des vertébrés, ce qui signifie qu’ils doivent, eux aussi, assurer des fonctions essentielles. Selon certaines hypothèses, ils spécifient la synthèse de protéines impliquées dans le contrôle de la croissance cellulaire, du développement embryonnaire et de la différenciation. Il est devenu clair, au cours des années quatre-vingt, que leur mutation, ou leur insertion près de sites qui activent leur expression sont fréquemment associées à l’apparition de cancers chez les animaux et chez l’homme. Vient à l’appui de cette façon de voir la découverte que le remplacement d’une seule base par une autre dans une séquence oncogène particulière transforme des cellules en culture en cellules cancéreuses, le même type de mutation étant décelé au niveau de la même séquence dans des tissus cancéreux humains. Cette mutation se traduit au niveau de la protéine synthétisée, dont la fonction est encore inconnue, par le remplacement d’un seul acide aminé par un autre, ce qui se répercute sur la conformation. Par ailleurs, dans certains cancers (comme le lymphome de Burkitt), la transformation maligne est très étroitement associée au transfert à la suite de la translocation d’une séquence oncogène d’un chromosome à un autre à proximité d’un site d’activation ce qui se traduit par un taux élevé de transcription et la synthèse d’une quantité accrue de la protéine concernée.Il est clair que de tels événements, même s’ils ne sont pas responsables à eux seuls des processus de cancérisation, n’en constituent pas moins une étape importante et sans doute le ou l’un des facteurs déclenchants. Et c’est un des plus récents succès de la biologie moléculaire que de les avoir mis en évidence.Comment cette relative instabilité de l’ADN s’accorde-t-elle avec ce que l’on sait du développement et de la différenciation au cours desquels des gènes s’expriment ou ne s’expriment plus de façon ordonnée et coordonnée? Des perspectives se dégagent. Il a été démontré par S. Tonegawa (Prix Nobel de médecine 1987) que lors de la différenciation des cellules immunocompétentes interviennent de profonds remaniements des gènes impliqués dans la synthèse des anticorps. Pourtant, le reste du génome ne semble pas être sujet à de telles modifications, non plus que le génome des autres lignées cellulaires. Existe-t-il chez ces dernières des systèmes de régulation qui inhibent l’activité des enzymes impliqués, est-ce la structure même de l’ADN qui est en cause? Quelles sont les lois qui président à ce qui apparaît pour l’heure appartenir au domaine de l’aléatoire?Autant de questions qui se posent, et pas seulement dans ce domaine. Les succès passés et présents de la biologie moléculaire, la finesse avec laquelle elle permet de disséquer le fonctionnement des êtres vivants donnent l’assurance qu’elle pourra fournir des réponses aux questions posées dans les différents thèmes qui viennent d’être évoqués et dans ceux qui n’ont pu l’être (mode d’action de certaines hormones, en particulier des hormones sexuelles, étude des mutants de développement, si importante pour notre compréhension de l’évolution au niveau macroscopique, architecture et fonctionnement du système nerveux...).
c’est au niveau de tels sites promoteurs que se fixent les répresseurs ou les activateurs de nature protéique identifiés chez les bactéries et qui commandent, en entrant ou non en compétition avec l’ARN polymérase le démarrage ou le non-démarrage de la synthèse de l’ARN concerné, autrement dit l’expression ou la mise au repos des gènes correspondants. On ne sait en revanche que peu de chose de la nature exacte des signaux nucléiques responsables de la fin d’une transcription, sinon que, là encore, il est vraisemblable qu’interviennent des protéines telles que celle qui a été isolée de E. coli : elle est nécessaire in vitro, dans un système de transcription reconstitué à la production de l’ARN de longueur normale tel qu’il est synthétisé in vivo; en son absence, les ARN produits sont anormalement longs.Élucider les mécanismes d’interactions entre séquences de bases nucléiques et protéines est d’une importance capitale pour la compréhension des phénomènes de régulation de l’expression des gènes. Car c’est ainsi que se pose le problème des mécanismes moléculaires qui fondent le déroulement des programmes de développement et de différenciation qui de l’œuf, ou de la graine, conduisent à la formation d’organismes pluricellulaires: certains gènes, ou groupes de gènes s’exprimant à certains stades précis du développement alors que d’autres sont mis «au repos».La structure fine de l’ADNCertaines méthodes physiques comme la diffraction des rayons X et des neutrons, l’utilisation du rayonnement synchrotron qui permet de réaliser des études tant dynamiques que structurales, les diverses techniques de la spectroscopie... fournissent déjà à cet égard des renseignements fort intéressants. Elles révèlent en particulier que ni les acides nucléiques ni les protéines ne sont des structures figées et que leurs interactions (ainsi qu’avec d’autres molécules) mettent en jeu des modifications de leur architecture, de leur conformation.Dans le même ordre d’idées, l’étude détaillée de la structure de la double hélice permet de montrer qu’elle n’est pas aussi régulière qu’on le pensait: elle présente de légères variations qui dépendent de la séquence des bases. Certaines de ces «irrégularités» sont reconnues par des protéines. On arrive ainsi à la notion qui se dégageait déjà de l’étude des protéines, selon laquelle la conformation de la molécule imposée par l’enchaînement de ses composants joue un rôle important pour ses propriétés biologiques. Cette notion se trouve confirmée par la mise en évidence d’enzymes qui modifient la topologie de la molécule, et par la mise en évidence de segments en hélice gauche alors que le modèle de Watson et Crick était en hélice droite, sur toute sa longueur. Enfin, il apparaît clairement que l’ADN est une structure dynamique sujette à des changements réversibles de conformation. Autant de données qui laissent prévoir des développements importants pour la connaissance des propriétés des macromolécules qui fondent leurs interactions.Identification des séquencesÀ partir des années soixante-dix, d’autres méthodes, de nouvelles techniques ont été élaborées qui ont fourni des résultats aussi intéressants que parfois inattendus. Citons parmi les principales:– La séparation des protéines par électrophorèse en double dimension donne la possibilité d’analyser qualitativement et quantitativement la plus grande partie sinon la totalité des protéines qui se trouvent ou qui sont synthétisées, par exemple, aux différents stades du développement d’un organisme donné; sa sensibilité est telle qu’elle peut révéler l’absence d’une seule protéine (parmi les milliers qu’elle contient) dans des cellules humaines mutantes ou des différences dans une seule protéine présente mais modifiée à la suite d’une mutation;– Les ARN messagers peuvent être isolés, purifiés, et leur traduction peut s’effectuer in vitro. Les protéines synthétisées dans ces conditions peuvent être comparées à celles qui le sont par des cellules intactes, ce qui fournit des renseignements sur les types de régulation qui opèrent au niveau de la traduction, au cours de la différenciation, par exemple;– L’hybridation moléculaire fondée sur la complémentarité des bases A-T (U); G-C. Elle fournit des renseignements précieux tant sur la structure des génomes quand il s’agit d’hybridation entre ADN et ADN, que sur les modalités de leur expression dans le cas des hybridations ADN-ARN. La méthode d’hybridation ADN-ADN a ainsi révélé qu’au contraire de ce qui se passe chez les bactéries, le génome des cellules eucaryotes est composite, formé de séquences semblables qui se trouvent répétées en de nombreux exemplaires et de séquences qui ne se trouvent représentées qu’une fois et dont font partie les gènes spécifiant la synthèse des protéines; cette même méthode permet d’isoler et de purifier les différentes classes de séquences; quant aux hybridations ADN-ARN, elles permettent, par exemple, de savoir avec quelle fréquence tel type de séquence d’ADN est transcrite, de déceler les espèces d’ARN spécifiques d’un état différencié donné, d’en mesurer les quantités synthétisées, de comparer entre eux les résultats obtenus sur différents types cellulaires depuis l’œuf, quand le matériel s’y prête, jusqu’aux états différenciés terminaux. C’est enfin l’hybridation moléculaire qui permet de repérer parmi les lignées bactériennes où se trouvent insérés les fragments d’ADN d’un autre organisme, celle qui porte le gène auquel on s’intéresse, le repérage se faisant par le biais d’une sonde radioactive (ARN ou ADN complémentaire, synthétisé à partir de ce dernier) qui s’hybride de façon sélective avec l’ADN recherché;– L’ADN peut être découpé en petits fragments grâce à des enzymes spécifiques, des endonucléases (ou enzymes de restriction) qui ont la propriété de scinder la molécule en des points précis de sa structure déterminés par la séquence de quelques nucléotides qui sont adjacents aux sites d’action des enzymes. Il est ainsi possible d’obtenir des fragments de petite taille que l’on peut isoler et caractériser, et d’établir une carte physique de la molécule par recoupement des fragments obtenus en faisant agir plusieurs nucléases qui diffèrent par la séquence des bases de leurs sites d’attachement. L’obtention de ces fragments courts rend possible l’analyse de la séquence des bases dont ils sont constitués. De nouvelles méthodes fiables et précises ont été élaborées qui permettent de déterminer la séquence exacte de plusieurs dizaines de milliers de bases.Isolement d’un gèneMais ces techniques n’auraient qu’un intérêt relativement limité s’il n’était pas possible d’avoir à l’état pur et en quantité suffisante le gène dont on veut étudier les propriétés. Or, chez un organisme pluricellulaire, tel que l’homme, par exemple, un gène donné ne représente que le millionième de l’ADN. D’où l’intérêt des techniques de clonage, base du génie génétique. Dans leur principe, elles consistent, après avoir fragmenté l’ADN, à insérer chacun des fragments dans l’ADN d’un vecteur (virus ou plasmide) capable de se répliquer dans une bactérie qui, en général, est E. coli . Le vecteur porte un gène qui confère la résistance des bactéries à un antibiotique. Le traitement de la population bactérienne par cet antibiotique aura pour effet de sélectionner celles qui sont résistantes du fait qu’elles hébergent bien l’ADN du vecteur lié au génome à étudier. Au pouvoir de multiplication du vecteur dans chaque cellule bactérienne, s’ajoute celui des bactéries elles-mêmes qui fournissent des populations aussi grandes qu’on le désire: le génome dont on veut étudier la structure et les propriétés se trouve donc amplifié de façon considérable. C’est ainsi que le génome humain, celui de la souris, de la drosophile... sont répartis dans différentes lignées, dans différents clones d’E. coli . Les fragmentations ayant été opérées de façon statistique (par voie enzymatique ou par voie mécanique), il y a une chance non négligeable pour que le gène auquel on s’intéresse soit en partie, en totalité (seul ou encore lié à ses segments adjacents), dans un ou quelques clones bactériens. Comment repérer ce ou ces clones? Soit par le produit de son expression... si le gène veut bien s’exprimer dans la bactérie soit, d’une façon plus générale, à l’aide d’une sonde. Celle-ci est constituée par l’ARN messager, rendu radioactif, qui spécifie la synthèse de la protéine qui est sous le contrôle du gène. Les techniques d’hybridation moléculaire permettent alors de repérer parmi tous les clones bactériens, le et seulement le clone qui contient le gène en question. Une variante de cette méthode consiste à utiliser comme sonde la molécule d’ADN radioactive synthétisée in vitro à partir de l’ARN messager pris comme matrice, à l’aide d’une enzyme, la transcriptase-réserve: on dispose ainsi d’une quantité importante de la molécule utilisée comme sonde, alors que l’ARN messager lui-même ne peut en général être obtenu qu’en petite quantité.Une fois le clone bactérien repéré, il suffira d’en extraire le vecteur et d’en libérer le gène en traitant ce vecteur par des enzymes de restriction.Les méthodes qui viennent d’être décrites, auxquelles il convient d’associer celles qui font appel à l’étude en microscopie électronique des molécules d’ADN, d’ARN et de leurs hybrides sont à l’origine d’une moisson de résultats dont certains exemples ont déjà été donnés (gènes éclatés, structure du gène de l’insuline (fig. 6), séquence des promoteurs...). Outre l’intérêt qu’elles présentent dans le domaine des biotechnologies (synthèse bactérienne d’insuline humaine ou d’autres hormones, de motifs antigéniques pour la production de nouveaux vaccins, étude des gènes qui contrôlent la fixation de l’azote chez les bactéries ou chez les symbiotes des plantes...), elles constituent un outil d’une finesse remarquable dans l’analyse des propriétés des êtres vivants.Contrôle des systèmes codantsOn en trouvera ici quelques exemples:D’abord, le fait de pouvoir isoler un gène avec ses parties adjacentes et d’en déterminer les séquences, permet d’accéder aux groupes de bases qui, situées à l’extérieur du gène proprement dit peuvent jouer le rôle de sites de reconnaissance pour les protéines régulatrices. Il est possible par voie chimique, de modifier directement et à volonté une base donnée, de faire par conséquent de la mutagenèse dirigée. L’étude des répercussions sur les propriétés fonctionnelles de la molécule permet de bien mieux les comprendre.Par ailleurs, connaissant la séquence des bases, il est possible d’en déduire la séquence des acides aminés de la protéine dont la synthèse est régie par le gène, méthode rapide et sûre quand il s’agit de protéines de poids moléculaire élevé dont l’analyse conventionnelle est difficile et qui fournit parfois des résultats intéressants en ce qu’elle révèle l’existence de séquences d’acides aminés qui seront éliminées lors de la transformation de la protéine synthétisée en protéine fonctionnelle. À l’inverse, connaissant la séquence de quelques acides aminés d’une protéine, et en déduisant la séquence des bases, on peut synthétiser par voie chimique la partie correspondante du gène. Celle-ci sera utilisée comme sonde: on évite ainsi l’isolement de l’ARN messager qui, sauf exception, est souvent une entreprise longue et délicate.Il est important, enfin, dans le cas des maladies héréditaires graves et invalidantes, et pour lesquelles aucun traitement n’est connu, de pouvoir prédire si l’enfant à naître présentera ou non cette maladie de façon, si c’est le cas, à pouvoir proposer un avortement aux parents. C’est le cas, par exemple, pour les anémies graves comme les thalassémies qui affectent la fonction de l’hémoglobine. Les gènes qui spécifient la synthèse des protéines constituants de cette molécule sont isolés. Utilisés comme sondes, ils ont permis de montrer que, dans certains cas, les troubles avaient pour origine la perte plus ou moins grande (parfois totale) du gène correspondant. L’étude d’ADN des cellules du fœtus, au moyen des enzymes de restriction permet de savoir si l’enfant à naître souffrira ou non de la maladie. La même méthodologie appliquée à l’anémie falciforme permet de tirer de semblables conclusions. Dans ce système des hémoglobines humaines, les études de génétique formelle et de génétique moléculaire aboutissent à dresser de façon précise la carte des gènes impliqués, montrant en particulier que les différents types d’hémoglobine apparaissent au cours du développement selon l’ordre dans lequel ces gènes sont situés les uns par rapport aux autres, et révélant l’existence de séquences en partie homologues qui ne sont pas exprimées.Instabilité du génomeL’une des découvertes les plus surprenantes concerne la relative instabilité du matériel génétique, aussi bien chez les procaryotes que chez les eucaryotes. Les modèles moléculaires d’ADN, ses photographies en microscopie électronique en donnent une image figée et statique qui ne correspond pas à la réalité. En fait, l’ADN est constamment soumis à des remaniements plus ou moins profonds: des gènes sont transposés d’un chromosome à un autre, des segments d’ADN sont insérés dans la molécule, d’autres en sont excisés, des recombinaisons s’effectuent, des lésions sont provoquées, qui sont réparées par des enzymes. On a donc affaire à un système qui se renouvelle. Quelques exemples permettront d’illustrer ce point.L’étude de trypanosomes, comme ceux qui sont à l’origine de la maladie du sommeil, avait montré que ces parasites échappent aux défenses immunitaires de l’hôte par le mécanisme suivant: lors de la première infection, l’hôte élabore des anticorps contre un déterminant antigénique du parasite. Celui-ci (ou une partie de la population) élabore alors rapidement un nouveau déterminant et le phénomène se reproduit. À ce jeu d’attaque et de réponse, l’hôte est perdant. D’où, en particulier, la difficulté de préparer des vaccins. Certaines des causes de cette diversité dans la production d’antigènes ont été élucidées lorsque les méthodes de la biologie moléculaire ont été appliquées à ce système. Le génome des trypanosomes contient une centaine d’exemplaires de séquences homologues mais qui diffèrent légèrement les unes des autres. Une seule de ces séquences, celle qui se trouve à proximité d’un site d’activation est exprimée à chaque fois, aboutissant à la synthèse d’un déterminant antigénique. En présence de l’anticorps formé contre ce déterminant, et par un mécanisme de transposition, la séquence qui s’exprimait est excisée et elle est remplacée par une copie de l’une de celles qui se trouvaient au repos et dont l’expression va se trouver déclenchée.Restant dans le domaine de l’immunologie, ce sont encore les méthodes de génie génétique qui ont permis de comprendre, au moins en grande partie, l’origine de l’extraordinaire diversité des anticorps. L’étude comparée de l’organisation des gènes concernés dans les cellules germinales et dans les cellules productrices d’anticorps révèle qu’au cours des processus de différenciation qui aboutissent à ces cellules compétentes (dont chacune n’exprime qu’un seul anticorps), de profonds remaniements interviennent. Ces remaniements se produisent au niveau de l’ADN, aboutissant à l’excision de séquences et à l’aboutage d’autres selon des combinaisons extrêmement diverses. À cela s’ajoutent, augmentant encore la diversité, des mutations somatiques et, au niveau, cette fois, des produits de transcription, de nouveaux remaniements qui sont caractéristiques des gènes en mosaïque. Un problème biologique fondamental se trouve ainsi éclairé d’un jour tout à fait nouveau.ÉvolutionUne des conséquences importantes de la mise en évidence de la relative instabilité de l’ADN concerne l’étude des mécanismes de l’évolution; un autre touche aux processus de différenciation.Pour évoluer, il est nécessaire de créer du nouveau, tout en restant viable et capable de transmettre la nouveauté à la descendance. Mutations de gènes et recombinaisons entre gènes étaient des mécanismes déjà connus des origines de la variabilité; de même que l’était le mode d’apparition de nouveaux gènes, copies formées à la suite d’accidents survenant lors de l’appariement des chromosomes homologues au cours de la méïose (crossing-over inégaux ou réparations à l’origine de la conversion, du retour d’une séquence mutée vers la séquence normale ou vice-versa). Les séquences des copies peuvent être modifiées tandis que les gènes d’origine continuent à assurer les fonctions habituelles de l’organisme. Si, à la suite de ces modifications, les nouveaux gènes confèrent, par exemple, un avantage aux organismes qui les portent, si elles se trouvent transposées à proximité de sites qui activent leur expression, elles pourront se répandre dans la population, être à l’origine de la manifestation de nouveaux caractères, de la mise en place de barrières génétiques entre les organismes parentaux et certains de leurs descendants, conduire à l’apparition de nouvelles espèces.Les arguments expérimentaux ne manquent pas, qui appuient ces conceptions et permettent d’aborder un aspect nouveau de la génétique des populations.Isoler un gène donné d’une espèce donnée, en déterminer la séquence, l’utiliser comme sonde chez les organismes appartenant à la même espèce, permet d’y déceler des séquences homologues légèrement modifiées, de mettre en évidence, par conséquent, un polymorphisme au niveau de l’ADN. Cela d’une façon plus directe et plus fiable que celle qui repose sur la comparaison de la séquence des acides aminés de protéines homologues et surtout en ayant accès à des séquences qui ne sont pas transcrites, comme les séquences régulatrices, ou qui, transcrites, ne sont pas traduites comme les introns. C’est ainsi qu’ont été mises en évidence des séquences proches de celles qui spécifient la synthèse des différentes globines qui entrent dans la composition des hémoglobines et qui, situées à proximité de celles-ci, ne s’expriment pas: les pseudo-gènes. C’est aussi ce type d’approche qui renforce la conception selon laquelle un gène ancestral unique serait à l’origine des différents gènes qui opèrent actuellement dans ce système.La même méthodologie permet de comparer les séquences de gènes homologues dans différentes espèces, de juger de leur conservation ou de leur vitesse d’évolution. Un exemple en a déjà été fourni, celui du gène de l’insuline humaine et d’un des gènes de l’insuline du rat: dans ce cas, les séquences ont peu évolué l’une par rapport à l’autre; en revanche l’autre gène de l’insuline du rat a évolué, lui, de façon indépendante et marquée. Dans le même ordre d’idées, les gènes des histones (protéines associées à l’ADN des cellules eucaryotes) et dont il existe de très nombreuses copies chez un même organisme, présentent un très haut degré d’homologie chez tous les organismes où ils existent, ce qui plaide en faveur de leur importance dans l’économie de la cellule. Le fait que ces séquences aient été conservées doit signifier que la structure des protéines spécifiées ne peut varier que dans de très étroites limites sous peine de voir leurs fonctions abolies.Différenciation - oncogenèseUn dernier exemple, et qui concerne tout autant les processus de différenciation, est celui des séquences oncogènes. Il s’agit de séquences de l’ADN cellulaire homologue aux gènes de virus qui ont la propriété de transformer des cellules normales en cellules cancéreuses. Ces gènes ont vu leurs séquences remarquablement bien conservées au cours de l’évolution des vertébrés, ce qui signifie qu’ils doivent, eux aussi, assurer des fonctions essentielles. Selon certaines hypothèses, ils spécifient la synthèse de protéines impliquées dans le contrôle de la croissance cellulaire, du développement embryonnaire et de la différenciation. Il est devenu clair, au cours des années quatre-vingt, que leur mutation, ou leur insertion près de sites qui activent leur expression sont fréquemment associées à l’apparition de cancers chez les animaux et chez l’homme. Vient à l’appui de cette façon de voir la découverte que le remplacement d’une seule base par une autre dans une séquence oncogène particulière transforme des cellules en culture en cellules cancéreuses, le même type de mutation étant décelé au niveau de la même séquence dans des tissus cancéreux humains. Cette mutation se traduit au niveau de la protéine synthétisée, dont la fonction est encore inconnue, par le remplacement d’un seul acide aminé par un autre, ce qui se répercute sur la conformation. Par ailleurs, dans certains cancers (comme le lymphome de Burkitt), la transformation maligne est très étroitement associée au transfert à la suite de la translocation d’une séquence oncogène d’un chromosome à un autre à proximité d’un site d’activation ce qui se traduit par un taux élevé de transcription et la synthèse d’une quantité accrue de la protéine concernée.Il est clair que de tels événements, même s’ils ne sont pas responsables à eux seuls des processus de cancérisation, n’en constituent pas moins une étape importante et sans doute le ou l’un des facteurs déclenchants. Et c’est un des plus récents succès de la biologie moléculaire que de les avoir mis en évidence.Comment cette relative instabilité de l’ADN s’accorde-t-elle avec ce que l’on sait du développement et de la différenciation au cours desquels des gènes s’expriment ou ne s’expriment plus de façon ordonnée et coordonnée? Des perspectives se dégagent. Il a été démontré par S. Tonegawa (Prix Nobel de médecine 1987) que lors de la différenciation des cellules immunocompétentes interviennent de profonds remaniements des gènes impliqués dans la synthèse des anticorps. Pourtant, le reste du génome ne semble pas être sujet à de telles modifications, non plus que le génome des autres lignées cellulaires. Existe-t-il chez ces dernières des systèmes de régulation qui inhibent l’activité des enzymes impliqués, est-ce la structure même de l’ADN qui est en cause? Quelles sont les lois qui président à ce qui apparaît pour l’heure appartenir au domaine de l’aléatoire?Autant de questions qui se posent, et pas seulement dans ce domaine. Les succès passés et présents de la biologie moléculaire, la finesse avec laquelle elle permet de disséquer le fonctionnement des êtres vivants donnent l’assurance qu’elle pourra fournir des réponses aux questions posées dans les différents thèmes qui viennent d’être évoqués et dans ceux qui n’ont pu l’être (mode d’action de certaines hormones, en particulier des hormones sexuelles, étude des mutants de développement, si importante pour notre compréhension de l’évolution au niveau macroscopique, architecture et fonctionnement du système nerveux...).
● Biologie moléculaire science consacrée à l'étude des molécules supportant le message héréditaire (acides nucléiques A.D.N. et A.R.N.). [La biologie moléculaire analyse, dans les molécules, la structure du génome et ses altérations (mutations) ainsi que les mécanismes de l'expression, normale et pathologique, des gènes. L'expression biologie moléculaire est parfois employée pour désigner les techniques d'étude des gènes.]
Encyclopédie Universelle. 2012.
